
引言
有些软件总能以某种形式劝退你,让你毫不犹豫地投入其他软件的怀抱,外加产生一种不吐不快的情愫,激发一种唯恐他人也会“遭此毒手”的正义感。软件之所以被称为软件,大抵是因为经过包装与测试,比较成熟且有很大的兼容性,至少按照文档来运行应该不会有问题,如果动不动就出错那与脚本何异?生信分析软件很大一部分都属于小众软件,有些软件也许本身的出发点就是科研与文章,而好不好用能不能让更多的人使用并不重要。遇到这样的软件,那就启用我们的万能躺平招式:分析软件千千万,不行咱就换一换。同时,对于这类软件应该保持敬畏之心,那句话怎么说来着:明知山有虎,猛敲退堂鼓。不然浪费的可是自己宝贵的时间,下面我们一起来看看今天的主角。
缘起
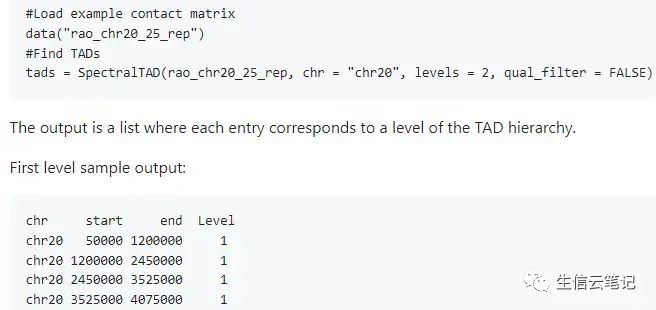
为什么会用到SpectralTAD呢?这都源自于做TAD差异分析时使用的TADCompare软件,关于该软件也写过两篇帖子:[],[]。TADCompare分析时可以接受预定的TAD文件,而软件文档中调用TAD使用的就是SpectralTAD,这便有了下面的事情。SpectralTAD是一个专门用于从HiC接触矩阵中进行TAD calling的R包,其使用一种基于滑动窗口的改进版光谱聚类方法来快速检测TAD。SpectralTAD将接触矩阵作为输入,并输出BED格式的数据框,其中包含TAD相对应的坐标位置。该工具包含SpectralTAD、SpectralTAD_Par两个函数,分别为单CPU和多CPU模式。另外,该包接受多种格式,如n × n、n × (n + 3)、3列等。至此,感觉SpectralTAD一切还挺正常,跟着文档代码走一遭才知道该软件非常难以驾驭,一起来看看怎么回事:
devtools::install_github("dozmorovlab/SpectralTAD")
library(SpectralTAD)
data("rao_chr20_25_rep")
tads = SpectralTAD(rao_chr20_25_rep, chr = "chr20", levels = 2, qual_filter = FALSE)
Converting to n x n matrix
Matrix dimensions: 2517x2517
Error in `vec_init()`:
! `x` must be a vector, not `NULL`.
Run `rlang::last_trace()` to see where the error occurred.
Warning messages:
1: Unknown or uninitialised column: `Group`.
2: Unknown or uninitialised column: `Group`.
else {
gap_order = order(-point_dist)
sil_score = c()
for (cluster in clusters) {
k = 1
partition_found = 0
first_run = TRUE
cutpoints = c()
while (partition_found == 0) {
new_gap = gap_order[k]
cutpoints = c(cutpoints, new_gap)
diff_points = which(abs(new_gap - cutpoints[-length(cutpoints)]) <=
min_size)
if (length(diff_points) > 0) {
cutpoints = cutpoints[-length(cutpoints)]
}
if (length(cutpoints) == cluster) {
partition_found = 1
}
else {
k = k + 1
}
}
if (any(is.na(cutpoints))) {
next
}
cutpoints = cutpoints[order(cutpoints)]
cutpoints = c(1, cutpoints, length(non_gaps_within) +
1)
group_size = diff(cutpoints)
memberships = c()
for (i in seq_len(length(group_size))) {
memberships = c(memberships, rep(i, times = group_size[i]))
}
sil = summary(cluster::silhouette(memberships,
dist_sub))
sil_score = c(sil_score, sil$si.summary[4])
Group_mem[[cluster]] = memberships
}
end_group = Group_mem[[which(diff(sil_score) < 0)[1]]]
if (length(end_group) == 0) {
end_group = dplyr::bind_rows()
}
else {
end_group = data.frame(ID = as.numeric(colnames(sub_filt)),
Group = end_group)
end_group = end_group %>% dplyr::mutate(group_place = Group) %>%
dplyr::group_by(group_place) %>% dplyr::mutate(Group = max(ID)) %>%
ungroup() %>% dplyr::select(ID, Group)
}
}
if (end == nrow(cont_mat)) {
Group_over = dplyr::bind_rows(Group_over, end_group)
end_loop = 1
}
else {
end_IDs = which(end_group$Group == last(end_group$Group))
start = end - length(end_IDs) + 1
if (length(start) == 0) {
start = end
}
end = start + window_size
end_group = end_group[-end_IDs, ]
Group_over = dplyr::bind_rows(Group_over, end_group)
if ((end + (2e+06/resolution)) > nrow(cont_mat)) {
end = nrow(cont_mat)
}
}
tads = SpectralTAD(rao_chr20_25_rep, chr = "chr20", levels = 2, qual_filter = FALSE, min_size=4)
Error in `vec_init()`:
! `x` must be a vector, not `NULL`.
Run `rlang::last_trace()` to see where the error occurred.
Warning messages:
1: Unknown or uninitialised column: `Group`.
2: Unknown or uninitialised column: `Group`.
tads = SpectralTAD(rao_chr20_25_rep, chr = "chr20", levels = 2, qual_filter = FALSE, min_size=3)
Error in `vec_init()`:
! `x` must be a vector, not `NULL`.
Run `rlang::last_trace()` to see where the error occurred.
Warning messages:
1: Unknown or uninitialised column: `Group`.
2: Unknown or uninitialised column: `Group`.
tads = SpectralTAD(rao_chr20_25_rep, chr = "chr20", levels = 2, qual_filter = FALSE, min_size=2)
head(tads$Level_1)
# A tibble: 6 × 4
chr start end Level
1 chr20 50000 325000 1
2 chr20 325000 525000 1
3 chr20 525000 775000 1
4 chr20 775000 1200000 1
5 chr20 1200000 1450000 1
6 chr20 1450000 1775000 1
自己测试:
这个错误来得突然,也让人有点摸不到头脑。怎么回事,这软件还挑人不成?当然,软件不可能因人而异,但会因环境而异。经过一番尝试,发现下面这一段代码:
运行文档中的代码之所以出错是因为endgroup没有结果,dplyr::bindrows(Groupover, endgroup)合并数据时引发了错误,也由此找到了相关联的参数min_size:
tads = SpectralTAD(rao_chr20_25_rep, chr = "chr20", levels = 2, qual_filter = FALSE, min_size=2, out_format='juicebox')
New names:
• `chr` -> `chr...1`
• `start` -> `start...2`
• `end` -> `end...3`
• `chr` -> `chr...4`
• `start` -> `start...5`
• `end` -> `end...6`
Error in `mutate()`:
ℹ In argument: `start = format(start, scientific = FALSE)`.
Caused by error:
! `start` must be size 1709 or 1, not 14.
当以为可以正常运行时,软件又告诉你没那么简单。帮助信息中虽然提示可以通过out_format参数修改输出格式:juicebox、bedpe、hicexplorer、bed,但是一运行又掉入另外一个坑:

算了,真心累了,不再折腾了,这个时候只想对软件说一句:就此别过,后会无期!
结语
分析时还是选择知名度好、出场率高的软件吧,毕竟这样的软件更加成熟,即使出了问题也能快速找到相应的解决办法,用不着花费大量的时间在软件上。除非没有更好的选择,否则还是不要啃硬骨头的好,不然时间浪费不说还可能消化不良,不如一开始就避而远之。
当然,不可否认,造成软件无法正常运行的原因很可能是与开发者的环境不同。所以,分析时使用软件更多的时候都是在与软件的环境和参数作斗争,躺不平的时候还是要支棱起来:生信路不平,还需要缝缝补补。
往期回顾
 量子哈希
量子哈希









![[讲堂]5公里跑进20分内 这些方法包教包会-量子哈希](https://www.ccwifi.cc/blogs/wp-content/uploads/2024/03/1710434074967_1.jpg)

